Goedkoop genoom
De kosten voor het bepalen van het menselijk genoom zakken richting duizend dollar. Daarmee is de grens nog niet bereikt. Binnen tien jaar zal DNA-sequencing gemeengoed zijn in de medische sector en de manier van diagnostiek en behandeling blijvend veranderen. ‘Dan komt niemand meer een ziekenhuis binnen zonder dat toestemming wordt gevraagd om mee te werken aan de ontcijfering van de genetische code.’
De magische grens van duizend dollar voor de ontcijfering van één volledig menselijk genoom is in zicht. De snelle ontwikkeling van DNA-sequencingtechnologie heeft verstrekkende gevolgen voor de wijze van diagnostiek en medische behandeling. ‘Over een jaar of tien zal het gemeengoed zijn dat iedereen bij opname in het ziekenhuis direct zijn volledige genoom laat ontcijferen. De kosten van DNA-sequencing dalen zelfs zo hard dat het in kaart brengen van onze genetische blauwdruk misschien wel een klusje voor de huisarts wordt’, zegt een enthousiaste prof.dr. Johan den Dunnen van het Leids Universitair Medisch Centrum. ‘DNA-sequencing gaat ons leven veranderen.’
‘DNA-sequencing gaat ons leven veranderen’
De stelligheid waarmee Den Dunnen de impact van zijn vakgebied beschrijft, zal menige wenkbrauw doen optrekken, maar gezien het extreem hoge tempo waarmee tal van bedrijven en onderzoekinstellingen nieuwe technieken ontwikkelen om DNA te ontcijferen, kan het hoofd van het Leids Genoom Technologie Centrum weleens gelijk krijgen. DNA-sequencing is in een door technologie gedreven stroomversnelling beland, vergelijkbaar met de ontwikkeling van de chip, die door de befaamde wet van Moore wordt beschreven.
 DNA-sequencing behelst het in kaart brengen van de genetische code van organismen. De twee strengen waaruit een DNA-molecuul is opgebouwd, zijn verbonden door basenparen. Er zijn vier verschillende basetypen, die worden aangeduid met de letters G, A, T en C. Het complete menselijke DNA ofwel genoom bestaat uit zo’n drie miljard basenparen. Deze omvangrijke code vormt een soort blauwdruk voor de ontwikkeling van een organisme en bepaalt in sterke mate de eigenschappen en het functioneren ervan. Uit biologisch en medisch oogpunt geldt het ontcijferen van DNA als een heilige graal, omdat het een fundamenteel inzicht in de werking van een organisme kan verschaffen en in de daarin aanwezige gebreken.
DNA-sequencing behelst het in kaart brengen van de genetische code van organismen. De twee strengen waaruit een DNA-molecuul is opgebouwd, zijn verbonden door basenparen. Er zijn vier verschillende basetypen, die worden aangeduid met de letters G, A, T en C. Het complete menselijke DNA ofwel genoom bestaat uit zo’n drie miljard basenparen. Deze omvangrijke code vormt een soort blauwdruk voor de ontwikkeling van een organisme en bepaalt in sterke mate de eigenschappen en het functioneren ervan. Uit biologisch en medisch oogpunt geldt het ontcijferen van DNA als een heilige graal, omdat het een fundamenteel inzicht in de werking van een organisme kan verschaffen en in de daarin aanwezige gebreken.
De eerste succesvolle sequencingmethoden, die begin jaren zeventig werden ontwikkeld, waren zeer complex, kostbaar en vooral tijdrovend. Ook bij aanvang van het ambitieuze Human Genome Project, dat in 1990 startte met als doel het menselijk genoom te ontcijferen, leverde een week werk hooguit enkele duizenden G’s, A’s, T’s en C’s op. Het duurde dan ook tot 2003 voordat het project werd afgerond. De presentatie van de drie miljard letters tellende menselijk bouwcode vormde een indrukwekkende wetenschappelijke mijlpaal. Het prijskaartje was eveneens indrukwekkend: drie miljard dollar.
Mijlpaal
Nog geen tien jaar na afronding van het Human Genome Project is een volgende mijlpaal bereikt: dit jaar presenteerde de Amerikaanse onderneming Life Technologies een sequencer die naar eigen zeggen een volledig genoom voor minder dan duizend dollar kan ontcijferen. De spectaculaire daling in de kosten gaat bovendien gepaard met een radicale reductie van de benodigde tijd. Het apparaat van Life Technologies, dat eerder lijkt op een laserprinter dan op een complex apparaat voor wetenschappelijk en medisch onderzoek, zou de drie miljard letters in minder dan één dag kunnen produceren. Andere fabrikanten claimen prestaties in dezelfde orde van kosten en tijd.
De momenteel meest gangbare DNA-sequencingmethoden halen deze presentaties nog niet. Ze worden weliswaar aangeduid als next generation sequencing, maar dat heeft vooral betrekking op de grotere capaciteit, ofwel de hoeveelheid basen die in één keer is af te lezen. Het ontcijferen van een genoom duurt echter nog steeds een paar dagen tot meer dan een week en kost enkele duizenden of tienduizenden euro’s. De methoden hebben een aantal stappen gemeen. Ten eerste werken ze allemaal met relatief korte DNA-fragmenten. Een compleet molecuul is eenvoudigweg te lang om met een hoge nauwkeurigheid uit te kunnen lezen. De strengen worden daarom opgeknipt in stukken van driehonderd tot duizend basenparen. Daarnaast maken bijna alle technieken gebruik van een amplificatiestap: ter verbetering van de nauwkeurigheid en vergroting van het uiteindelijke signaal is het noodzakelijk de fragmenten te vermenigvuldigen. Dit gebeurt doorgaans via een met behulp van bacteriën ingezette speciale kettingreactie. De derde stap is recombinatie: de ontcijferde fragmenten worden met complexe computeralgoritmes weer aan elkaar geplakt. De methoden en systemen verschillen vooral van elkaar ten aanzien van de mate van integratie en uiteindelijke detectie van de G’s, A’s, T’s en C’s.
‘Vermenigvuldigingsfouten zijn lastig te voorkomen’
Het Amerikaanse biotechnologiebedrijf 454 Life Sciences, dat in de top vijf van sequencerfabrikanten staat, brengt apparaten op de markt op basis van een methode genaamd pyrosequencing. Het bedrijf maakt gebruik van minuscule balletjes waarop een groot aantal kleine stukjes DNA zitten. Deze vormen elk een adapter, waaraan een te ontcijferen DNA-fragment zich kan vasthechten. Aan elk balletje wordt precies één fragment geplakt. Vervolgens wordt dat fragment vermenigvuldigd, zodat op de andere adapters van het balletje identieke DNA-fragmenten van enkele honderden basen lang groeien. Na de vermenigvuldigingsstap wordt een van de twee DNA-strengen van de fragmenten door middel van een temperatuurverhoging verwijderd. Per balletje resteert een set enkelvoudige DNA-strengen. Op deze manier ontstaan miljoenen balletjes voorzien van enkelvoudige DNA-strengen.
‘Het eigenlijke pyrosequencen vindt vervolgens plaats in een dun plaatje, dat niet veel groter is dan een creditcard’, vertelt Todd Arnold, vicepresident R&D bij 454 Life Sciences. Het plaatje bevat 1,7 miljoen putjes van ongeveer 29 μm in diameter. In elk putje wordt één balletje met DNA-fragementen geplaatst, waarna dat wordt opgevuld met een mix van onder meer DNA-polymerase, dat ontbrekende basen aan de streng kan plakken, en een bioluminescent enzym. ‘Hierna worden oplossingen met steeds één basetype over het plaatje gespoeld. Incorporatie van zo’n nucleotide resulteert in een kleine lichtpuls. Boven het plaatje bevindt zich een extreem gevoelige camera, die de lichtpulsjes uit de verschillende putjes detecteert. Door de pulsjes te relateren aan het aanwezige basetype kan de volledige trits G’s, A’s, T’s en C’s per fragment worden bepaald, want alleen de letterscombinaties AT, TA, GC en CG zijn mogelijk.’
‘De forse kostendaling resulteert in een brede introductie van de technologie’
Een groot voordeel van de methode van 454 Life Sciences zijn de lange DNA-fragmenten waarmee de systemen overweg kunnen. ‘Momenteel is per balletje een lengte tot zevenhonderd basen haalbaar’, stelt Arnold. De grote lengte vergemakkelijkt onder meer het weer op volgorde leggen van de fragmenten. Ook in termen van nauwkeurigheid presteert pyrosequencing goed. De onderneming claimt een nauwkeurigheid van 99,9 %. Alleen de kosten zijn fors: het ontcijferen van een miljoen basen kost ongeveer tien dollar. De apparaten van het eveneens Amerikaanse bedrijf Illumina produceren eenzelfde verzameling data voor slechts tien dollarcent. De techniek is enigszins vergelijkbaar. Illumina laat DNA-fragmenten zich direct aan een glasplaatje hechten en zich ter plaatse vermenigvuldigen. Daardoor ontstaan clusters van identieke fragmenten. Deze worden vervolgens blootgesteld aan een oplossing met daarin vier typen speciale basen, die zich als een label aan een groeiende streng binden. Deze zijn fluorescent en hierdoor detecteerbaar met een camera. Het lezen van een complete set clusters neemt met enkele dagen relatief veel tijd in beslag, maar doordat er heel veel clusters op een plaatje passen, levert één run wel veel data op. Illumina claimt in één keer tot driehonderd miljard basen af te kunnen lezen. De nauwkeurigheid is vergelijkbaar met de methode van 454 Life Sciences, maar de kleine fragmentlengte van 250 basen beperkt de mogelijkheden om alle stukken weer op volgorde te leggen.
 Terwijl de sequencers van Illumina en 454 Life Sciences momenteel op grote schaal worden ingezet en ze goede prestaties leveren, dient zich alweer een nieuwe generatie machines aan. ‘Fabrikanten zetten momenteel twee belangrijke stappen voorwaarts’, vertelt Den Dunnen. ‘In de eerste plaats komen er sequencingmethoden op de markt waarbij niet langer labels zijn vereist.’ Bijna alle nu gangbare technieken gebruiken fluorescerende labels om de inbouw van DNA-basen te meten. Deze labels en de benodigde hulpstoffen zijn echter duur en verantwoordelijk voor een fors deel van de operationele kosten. Omdat DNA-sequening met behulp van labels doorgaans extreem gevoelige camera’s en optische elementen vergt, hangt ook aan de machines een fors prijskaartje. ‘De camera’s zijn enorm duur. Bovendien maakt het gebruik van optica de fabrikanten van sequencingapparatuur afhankelijk van de producenten van beeldchips. De ontwikkeling hiervan laat zich niet sturen door de behoefte aan betere DNA-sequencers.’
Terwijl de sequencers van Illumina en 454 Life Sciences momenteel op grote schaal worden ingezet en ze goede prestaties leveren, dient zich alweer een nieuwe generatie machines aan. ‘Fabrikanten zetten momenteel twee belangrijke stappen voorwaarts’, vertelt Den Dunnen. ‘In de eerste plaats komen er sequencingmethoden op de markt waarbij niet langer labels zijn vereist.’ Bijna alle nu gangbare technieken gebruiken fluorescerende labels om de inbouw van DNA-basen te meten. Deze labels en de benodigde hulpstoffen zijn echter duur en verantwoordelijk voor een fors deel van de operationele kosten. Omdat DNA-sequening met behulp van labels doorgaans extreem gevoelige camera’s en optische elementen vergt, hangt ook aan de machines een fors prijskaartje. ‘De camera’s zijn enorm duur. Bovendien maakt het gebruik van optica de fabrikanten van sequencingapparatuur afhankelijk van de producenten van beeldchips. De ontwikkeling hiervan laat zich niet sturen door de behoefte aan betere DNA-sequencers.’
De Amerikaanse onderneming Ion Torrent, een onderdeel van het concern Life Technologies, presenteerde twee jaar terug het eerste labelvrije sequencingsysteem. De methode toont overeenkomsten met de techniek van 454 Life Sciences. Ion Torrent gebruikt eveneens kleine balletjes waar enkelvoudige DNA-strengen zich aan hechten, waarna de stukjes worden vermenigvuldigd. Ook maakt Ion Torrent gebruik van een chip voorzien van kleine putjes, waarin de balletjes worden geplaatst. Ten slotte worden de putjes net als bij 454 Life Sciences tijdens het sequencen gevuld met een oplossing die afwisselend G’s, A’s, T’s of C’s bevat. Maar hier houden de overeenkomsten op.
Stroompjes
Ion Torrent benut een natuurlijk verschijnsel dat zich voordoet wanneer er een passende base aan een DNA-streng wordt gekoppeld. Bij de inbouw van een G, A, T of C komt er een waterstofion vrij. De chip met putjes is aan de onderzijde voorzien van een elektrisch circuit, dat deze positief geladen deeltjes in de vorm van kleine stroompjes detecteert. Door de pulsjes te relateren aan het op dat moment aanwezig basetype, kunnen de fragmenten per putje worden ontcijferd.
De presentatie van dit nieuwste sequencermodel eerder dit jaar deed veel stof opwaaien. De Ion Proton-machine zou namelijk binnen 24 uur een volledig menselijk genoom voor minder dan 1000 dollar kunnen ontcijferen. Het apparaat zelf is met een prijskaartje van zo’n 150 000 dollar bovendien een factor drie goedkoper dan apparatuur van concurrenten. ‘Het gebruik van chiptechnologie heeft als voordeel dat het zich laat opschalen zonder dat het sequencen meer tijd in beslag neemt’, stelt dr. Andrew Felton, directeur Productmarketing bij Ion Torrent. Een nadeel van deze technologie is de beperkte lengte van de DNA-fragmenten. ‘Lange strengen kunnen interacties met elkaar aangaan, wat de nauwkeurigheid negatief beïnvloedt’, legt Felton uit. De maximale fragmentlengte bedraagt momenteel zo’n tweehonderd baseparen.
‘We zullen eraan gewend raken dat we weten waarop we een verhoogd risico hebben’
De methode van Ion Torrent heeft met de andere technieken een relatief zwak element gemeen: de amplificatiestap. Hoewel die stap een goedkope routineklus is, bestaat het risico dat er bij de vermenigvuldiging van een DNAfragment kopieerfouten insluipen. ‘Amplificatiefouten zijn lastig te voorkomen en laten zich bij het sequencen slecht onderscheiden’, zegt Den Dunnen. ‘Idealiter wordt ook de amplificatiestap geëlimineerd en heeft een sequencer voldoende aan één DNAmolecuul. Dat is de tweede grote stap die momenteel wordt gezet: single molecule sequencing.’ Pacific Biosciences, de meest succesvolle aanbieder van single molecule sequencers, ontcijfert DNA-fragmenten met behulp van een plaatje dat is voorzien van minuscule golfpijpjes. Dit zijn 100 nm diepe cilindervormige gaatjes van zo’n 70 nm in doorsnee. In elk golfpijpje zitten één enkelvoudige DNAstreng en een oplossing met de vier basetypen. Aan deze basen zijn fluorescente moleculen bevestigd; elk type heeft een eigen kleur. De gaatjes bevatten tevens een polymerase, dat een kopie maakt van het aanwezige DNA-molecuul en steeds de passende base aan het fragment toevoegt. Bij de incorporatie van een letter komt het fluorescente molecuul los. Op het moment dat de letter wordt ingebouwd, is het korte tijd detecteerbaar met een digitale camera. Het aflezen gaat zeer snel: per seconde worden er twee of drie letters geregistreerd. De totale productie van een systeem is echter met zo’n honderd miljoen letters per uur beperkt.
‘Binnen tien jaar is sequencing gemeengoed in de medische sector’
Hoewel de apparatuur van Pacific Biosciences nog relatief duur is, liggen er tal van nieuwe toepassingen in het verschiet. Kwalitatief slechte DNA-fragmenten, die zich moeilijk laten vermenigvuldigen, kunnen met single molecule sequencing gemakkelijker worden ontcijferd. Bij forensisch onderzoek, waar rechercheurs niet altijd de luxe van een vers DNA-monster hebben, kan de techniek zodoende voordelen bieden. ‘Prenataal bloedonderzoek geldt als een andere veelbelovende toepassing’, zegt Den Dunnen. ‘Het bloed van de moeder bevat tijdens de zwangerschap stukjes uiteenvallend DNA van de foetus. Sequencing maakt het mogelijk het DNA van de ongeborene te ontcijferen en te scannen op eventuele genetische afwijkingen. Bloedafname bij de moeder kan dan een vruchtwaterpunctie vervangen.’ Vooral vanuit kostenoogpunt en snelheid biedt single molecule sequencing voordelen.
USB-stick
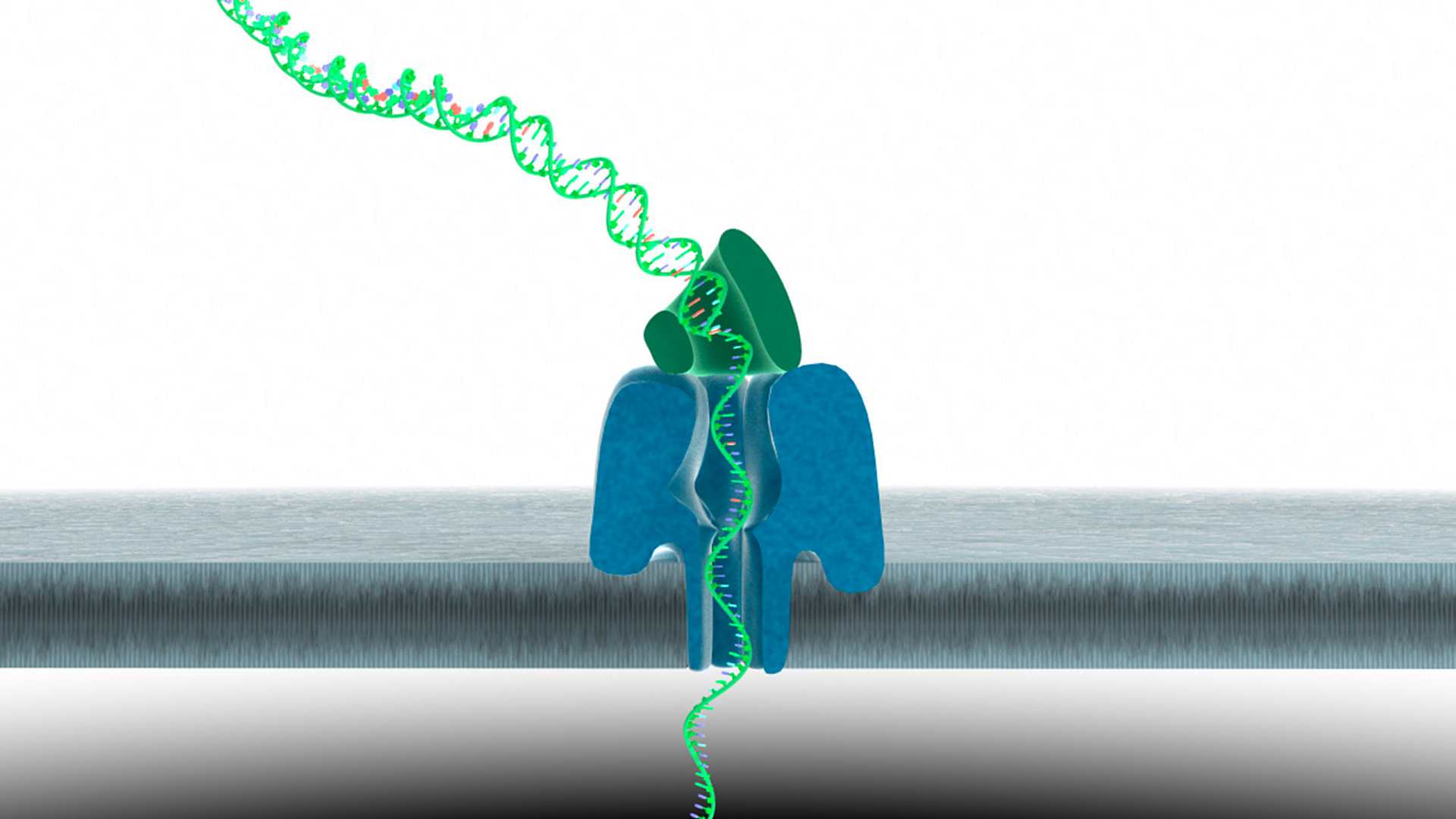
De eerste machines die zowel zonder labels werken als voldoende hebben aan één DNA-molecuul, zijn ook al in ontwikkeling. Ze moeten de voordelen van single molecule sequencing combineren met de lage kosten van labelvrije technologie. Alle ogen zijn hierbij gevestigd op Oxford Nanopore. Deze Britse onderneming kondigde begin dit jaar twee apparaten aan, die waarschijnlijk voor eind december op de markt komen. Het kleinste exemplaar heeft het formaat van een forse USB-stick en past ook in de USB-poort van een conventionele computer.
Hoewel het bedrijf nog terughoudend is met het verstrekken van details, is het concept wel al uit de doeken gedaan. Het uitgangspunt vormt een elektrisch isolerend membraan, zoals siliciumnitride, waarin met behulp van speciale eiwitten of synthetische stoffen een extreem kleine porie wordt gemaakt. Over het membraan wordt vervolgens een spanning aangebracht, waarbij er door de porie een ionische stroom gaat lopen. Deze stroom maakt het mogelijk een DNA-streng door het gaatje te trekken. Wanneer een base de porie passeert, vergroot dit de weerstand, waardoor de ionische stroom tijdelijk in sterkte afneemt. Deze variatie in de stroom wordt gebruikt om de genetische code te ontcijferen. De verschillende basetypen G, A, T en C zorgen elk namelijk voor een specifieke afname van de stroom. Door een extreem nauwkeurige meting van de fluctuaties is de volgorde te bepalen.
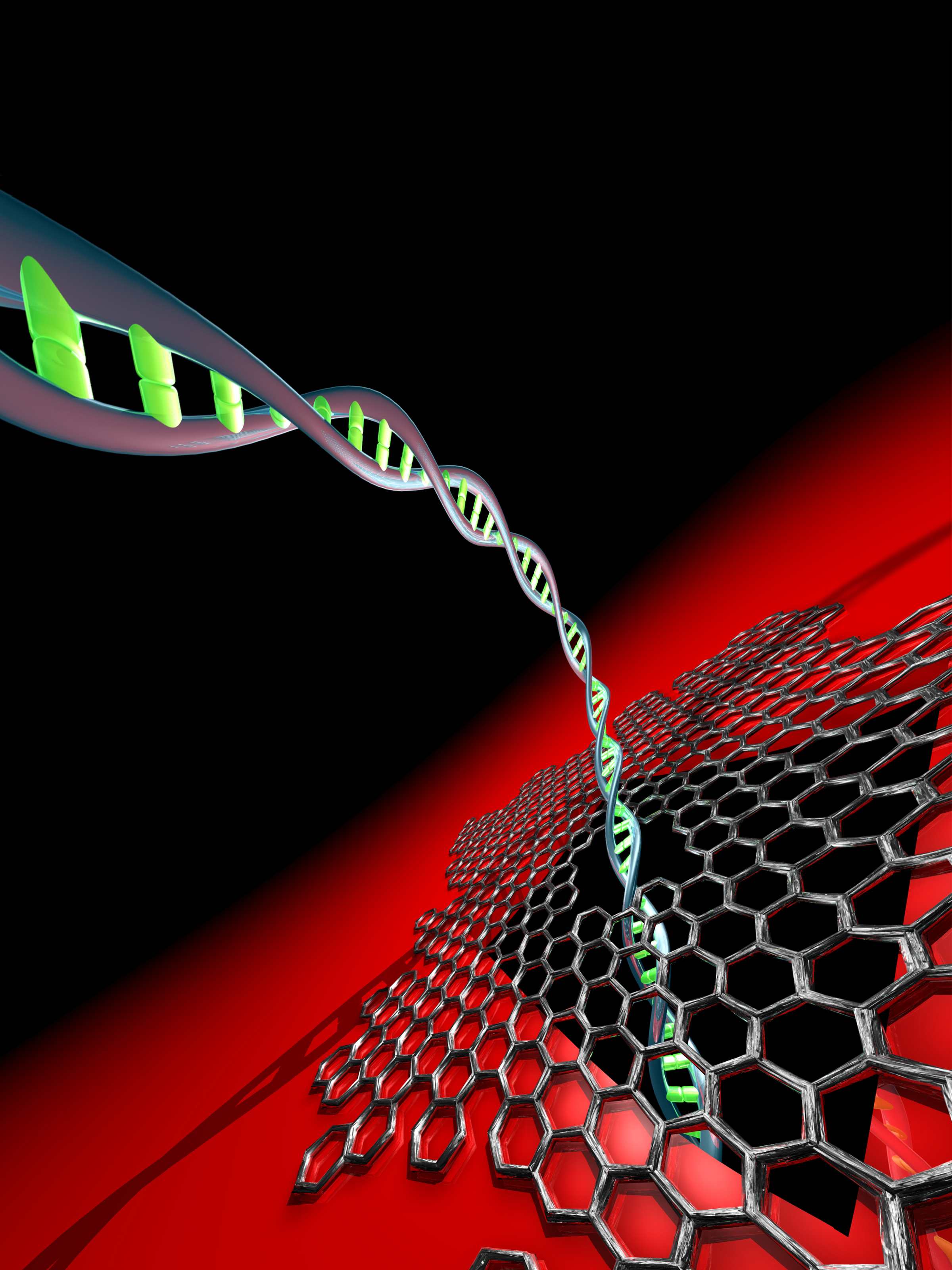 De techniek moet zich nog bewijzen, maar biedt wel veel potentie, meent prof. dr. Cees Dekker, hoogleraar Moleculaire Biofysica aan de TU Delft. Zijn groep verricht zelf onderzoek naar het gebruik van nanoporiën bij het ontcijferen van DNA. ‘Momenteel doen we onderzoek naar het gebruik van grafeen als membraan. De gebruikelijke membranen zijn zo’n 20 nm dik. DNA-basen zijn aanzienlijk kleiner, zodat er meerdere letters gelijktijdig in de porie zitten. Grafeen kan dit probleem oplossen, omdat het slechts één atoomlaag, ofwel 0,3 nm dik is.’ ‘Een andere moeilijkheid is de snelheid waarmee een DNAstreng door een porie gaat’, vervolgt Dekker. ‘Na het aanbrengen van een spanning flitst een streng er in ijltempo doorheen. Elke base bevindt zich misschien slechts enkele tientallen nanoseconden in de porie, wat het uitlezen van het signaal vrijwel onmogelijk maakt. Een optische pincet, die met behulp van lasers een kracht op een DNA-streng uitoefent, biedt mogelijk uitkomst. Hiermee is een streng gecontroleerd en langzamer door een porie te trekken. Onze groep onderzoekt momenteel een aantal technieken om dit voor elkaar te krijgen.’
De techniek moet zich nog bewijzen, maar biedt wel veel potentie, meent prof. dr. Cees Dekker, hoogleraar Moleculaire Biofysica aan de TU Delft. Zijn groep verricht zelf onderzoek naar het gebruik van nanoporiën bij het ontcijferen van DNA. ‘Momenteel doen we onderzoek naar het gebruik van grafeen als membraan. De gebruikelijke membranen zijn zo’n 20 nm dik. DNA-basen zijn aanzienlijk kleiner, zodat er meerdere letters gelijktijdig in de porie zitten. Grafeen kan dit probleem oplossen, omdat het slechts één atoomlaag, ofwel 0,3 nm dik is.’ ‘Een andere moeilijkheid is de snelheid waarmee een DNAstreng door een porie gaat’, vervolgt Dekker. ‘Na het aanbrengen van een spanning flitst een streng er in ijltempo doorheen. Elke base bevindt zich misschien slechts enkele tientallen nanoseconden in de porie, wat het uitlezen van het signaal vrijwel onmogelijk maakt. Een optische pincet, die met behulp van lasers een kracht op een DNA-streng uitoefent, biedt mogelijk uitkomst. Hiermee is een streng gecontroleerd en langzamer door een porie te trekken. Onze groep onderzoekt momenteel een aantal technieken om dit voor elkaar te krijgen.’
De onderzoeksgroep van Den Dunnen hoopt in de loop van volgend jaar een sequencer van Oxford Nanopore aan te schaffen. ‘Het kostenvoordeel zal de introductie van de nieuwe generatie DNA-sequencers bevorderen’, denkt Den Dunnen. ‘De forse kostendaling zal bovendien resulteren in een brede introductie van de technologie. Zelfs met het huidige prijsniveau van duizend dollar is het lonend om het volledige genoom van mensen te ontcijferen. Die investering wordt uiteindelijk ruimschoots terugverdiend. Neem medicatie als voorbeeld. Nu gebeurt het regelmatig dat mensen verkeerd reageren op een bepaald medicijn. Hiermee zijn enorme kosten gemoeid. Op basis van genetisch onderzoek is het gedeeltelijk mogelijk vooraf te bepalen hoe iemand op een bepaald medicijn reageert.’
‘Binnen tien jaar is DNA-sequencing gemeengoed in de medische sector’, voorspelt Den Dunnen. ‘Dan komt niemand een ziekenhuis meer binnen zonder dat toestemming wordt gevraagd om mee te werken aan de ontcijfering van de genetische code.’ Voordat het zover is, moet er wel een brede maatschappelijke discussie op gang komen. Willen mensen wel weten op welke aandoeningen ze een verhoogd risico hebben? Hoe worden DNA-gegevens afgeschermd? ‘Het zal een kwestie van wennen zijn. Maar na verloop van tijd raken we eraan gewend dat we weten waarop we een verhoogd risico hebben.’
Er ligt echter nog veel werk in het verschiet, niet alleen in de ontwikkeling van nog betere en goedkopere sequencers, maar ook in het opbouwen van kennis over het menselijk genoom. ‘Het kunnen ontcijferen van DNA is één ding; de vervolgvraag is wat we met al deze data gaan doen’, zegt drs. Bas Reichert, directeur en eigenaar van het Nederlandse BaseClear. Het bedrijf heeft een laboratorium vol met sequencers en werkt in opdracht van onderzoeksinstellingen en bedrijfsleven. ‘Van 95 % van het menselijk DNA is nog niet bekend wat het doet.’ De opkomst van DNA-sequencing opent zodoende compleet nieuwe deuren. Den Dunnen: ‘Dit gaat de wereld blijvend veranderen. De medische wereld heeft zich altijd een beetje moeten behelpen: repareren zonder de constructie te kennen. Straks kunnen medici gericht te werk gaan.’
Dit artikel van Thijs van Velzen staat in De Ingenieur van 19 november 2012






